Les gouvernements, les scientifiques et les instituts de recherche utilisent la technologie et l’innovation pour lutter contre le COVID-19.
Des outils open source ont été développés et certains continuent d’être testés pour soutenir les efforts qui ont été déployés pour minimiser les conséquences de ce virus mortel.
Une gamme d’outils open source est déjà en place, aidant les chercheurs à se renseigner sur la maladie, à identifier la propagation, à prévenir la propagation et à minimiser la propagation et les décès. Une gamme de projets est disponible au Écosystème de l’éducation qui explique comment utiliser différents outils.
Cet article examine certains des outils et bibliothèques open source qui sont utilisés dans la surveillance, la prévention et le confinement, le diagnostic et le traitement du COVID-19.
Table des matières
Carte de suivi COVID-19
En janvier, le Centre pour la science et l’ingénierie des systèmes du JHU a lancé un Carte de suivi mondiale COVID-19 qui est rapidement devenue une source internationalement fiable de données en temps réel sur l’évolution de la pandémie.
La carte est open source et a été utilisée pour alimenter les efforts de recherche et de visualisation par les principaux médias, les petites organisations et les gouvernements locaux.
DXY-COVID-19-Crawler
DXY-COVID-19-Crawler est l’un des premiers projets open source développés pour répondre à COVID-19 en janvier.
Les développeurs ont utilisé les données de DXY.cn, un site utilisé par les médecins chinois pour signaler et suivre les cas. Ils ont développé un robot d’exploration Web qui collectait les données du site et les rendait disponibles via une API et un entrepôt de données. Le code est disponible sur GithubGenericName.
Trousse de données ouvertes
Kit de données ouvertes (ODK) n’est pas un nouvel outil. Il a déjà été utilisé lors des épidémies d’Ebola en Afrique de l’Ouest en 2004 et de l’épidémie plus récente en République démocratique du Congo en 2019.
Il aide principalement les utilisateurs à collecter, gérer et utiliser les données dans la recherche des contacts, la cartographie stratégique, l’aide à la décision, la sensibilisation de la communauté et la gestion des cas.
La communauté ODK a encore avancé son utilisation en réponse au COVID-19 en mettant à disposition un formulaire de signalement dans ses déploiements. Le formulaire est conçu selon le protocole de l’Organisation mondiale de la santé pour enquêter sur les cas et rechercher les contacts et les enquêtes.
Les développeurs principaux offrent également une assistance gratuite à toute organisation qui a déployé l’ODK en réponse à COVID-19.
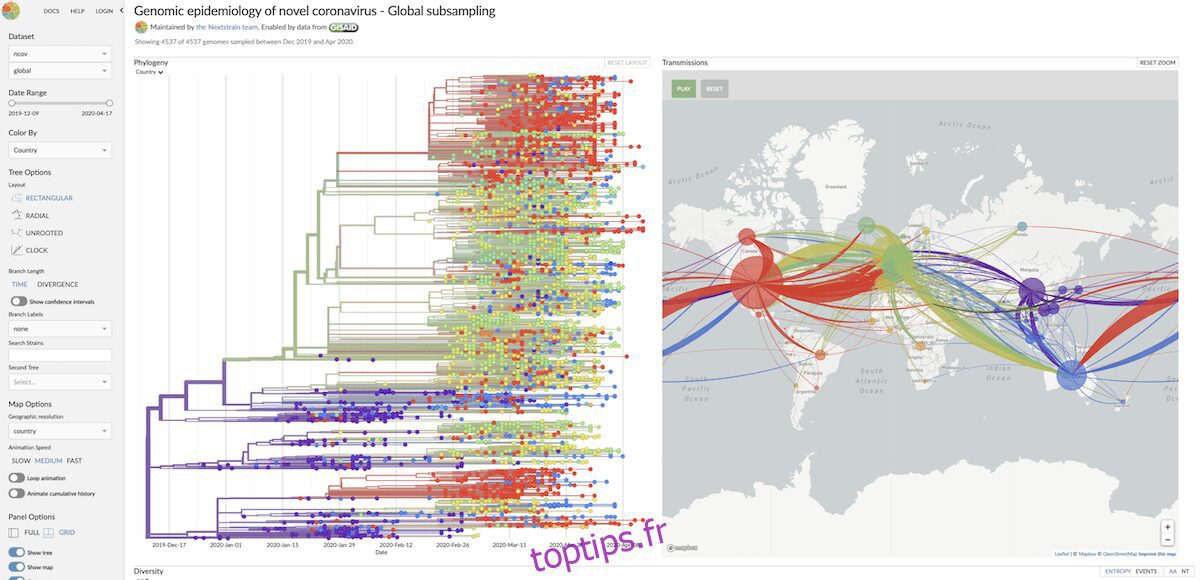
Prochaine souche
Prochaine souche suit l’évolution des agents pathogènes. Il a déjà été utilisé pour établir les antécédents familiaux d’une maladie, permettant ainsi de prédire la progression de la maladie.
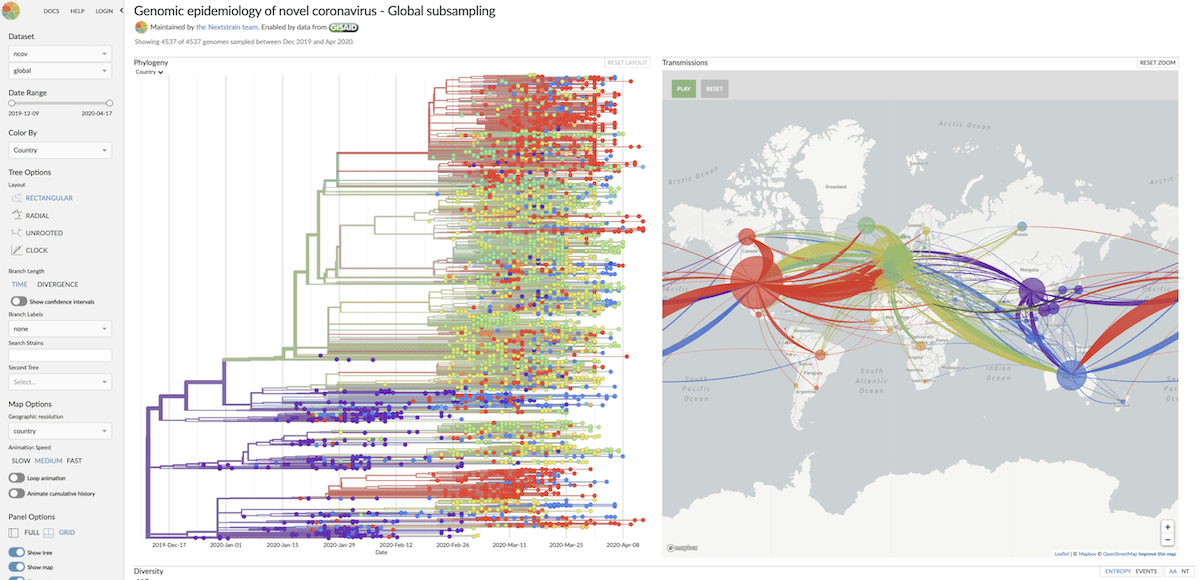
Il a été utilisé avec succès lors d’épidémies précédentes, comme Ebola. Grâce aux données génétiques de l’Initiative mondiale sur le partage de toutes les données sur la grippe (Gisaid), Nextxtrain est utilisé pour lutter contre le COVID-19.
DHIS2
Vous savez probablement déjà DHIS2. Il s’agit du plus grand système de gestion de l’information sur la santé au monde. Il est utilisé dans plus de 70 pays. Dans le cadre de la réponse au COVID-19, le DHIS2 a publié un ensemble de données numériques qui accélère la détection des infections, la notification, la surveillance et le traitement de la maladie.

Le paquet de données numériques DHIS2 utilise des métadonnées standard alignées sur le protocole de l’OMS sur la définition et la surveillance des cas de COVID-19 pour permettre un déploiement et une réponse rapides.
Pikobar Java occidental
Le Centre indonésien d’information et de coordination pour les maladies et les catastrophes est un centre de réponse aux crises formé pour atténuer et répondre au COVID-19 dans la province de Java occidental en Indonésie.
Le service numérique Jabar a, dans le cadre de la réponse, développé un outil et application Web open source qui permet aux utilisateurs d’accéder aux dernières données COVID-19.
OpenMRS
OpenMRS est un système de soins aux patients utilisé dans de nombreux pays en développement à travers le monde. En raison de la flexibilité du Système OpenMRSles pays dont les ressources en soins de santé ont été mises à rude épreuve peuvent l’utiliser pour la surveillance, le dépistage et le traitement du COVID-19.
Le système peut les aider à développer leurs capacités en leur donnant accès à des informations scientifiques sur la manière de faire face à la crise.
OpenLMIS
La Projet OpenLMIS utilise une tactique axée sur la communauté pour développer un système d’information de gestion logistique open source et adaptable. Le système OpenLMIS vise à améliorer l’exactitude des données, à accroître la responsabilité, à améliorer l’actualité des données et la visibilité.
Le système OpenLMIS vise à améliorer les chaînes d’approvisionnement en santé, l’inventaire des ressources médicales pour fournir une image claire des fournitures médicales disponibles, y compris les kits de test, et les EPI (équipements de protection individuelle). Cet outil peut être déployé efficacement pour aider les décideurs à allouer des ressources en réponse au COVID-19.
Sites de santé
La Projet de cartographie des sites de santé mondiaux est un projet visant à cartographier chaque établissement de santé dans le monde et à rendre les détails de chaque hôpital facilement accessibles. Les données sur les établissements de santé ont été rendues accessibles via une API.
En collaborant avec les utilisateurs, l’équipe de healthsites.io capture et valide l’emplacement et les coordonnées de chaque établissement de santé et rend les données librement disponibles et accessibles via une licence de données ouvertes.
SORMAS
SORMAS (Système de gestion et d’analyse de la réponse aux épidémies de surveillance) est un système de e-santé mobile open-source. Il a été déployé pour mettre en œuvre le contrôle des maladies ainsi que les procédures de gestion des épidémies.
Il a été efficacement déployé dans plusieurs pays, dont le Ghana, le Nigéria, le Népal et les Fidji, pour la surveillance et la détection précoce du COVID-19.
SORMAS est un système open source gratuit qui respecte les normes de protection des données.
COVID-19 à Tokyo
Contrairement à beaucoup d’autres villes et gouvernements, le Gouvernement métropolitain de Tokyo a développé un site Web open source qui informe ses résidents sur le COVID-19. En le rendant open-source, le site a vu les contributions de plus de 200 utilisateurs. Trois autres villes, Chiba, Nagano et Fukuoka, ont recréé le site Web.
OpenELIS
Le but de la Système de santé OpenELIS est d’améliorer les soins de santé en fournissant un système de gestion des informations de laboratoire progressif et basé sur des normes qui peut être utilisé par diverses initiatives de santé pour améliorer les options de traitement.
L’épidémie de COVID-19 a présenté un défi mondial de recherche des contacts et de dépistage de masse des cas suspects. Le système OpenELIS peut être efficacement déployé dans la lutte contre le COVID-19 pour faciliter le suivi des tests de laboratoire et des résultats.
La Boîte à outils de santé communautaire est une collection d’outils open source ainsi que de ressources en libre accès qui visent à créer et à déployer des outils numériques à utiliser dans les initiatives de santé communautaire dans les zones difficiles d’accès.
La communauté des développeurs de Community Health Toolkit s’est mobilisée pour développer des outils et des ressources visant à aider les agents de santé communautaires à lutter contre le COVID-19.
CARILLON
La Modèle d’impact hospitalier COVID-19 pour les épidémies (CHIME) est une application open source développée par des data scientists de Penn Medicine – Université de Pennsylvanie. Il s’agit d’un outil en ligne qui permet aux hôpitaux de prédire l’impact du virus sur les ressources de santé.
Il est développé à l’aide de Python et de la dépendance open source pandas.
Carte des soins COVID
La Carte des soins COVID aide à cartographier les ressources de soins de santé déjà existantes et à prévoir les lacunes dans les lits d’hôpitaux, les ventilateurs, les fournitures médicales et le personnel. Toutes les méthodes, outils de traitement de données, visualisations et code source sont gratuits et open-source.
Le projet COVID Care Map cherche à anticiper et à agir pour mobiliser du soutien afin de soigner efficacement le nombre croissant d’infections au COVID-19 et de personnes nécessitant des soins intensifs.
Locale.ai
Locale.ai a développé une visualisation interactive open-source de tous les cas confirmés de COVID-19 à travers le monde. Il interroge l’ensemble de données open source de l’Université John Hopkins.
Locale.ai a développé le Site de visualisation COVID-19 par l’utilisation de Vue.js, qui est un framework populaire qui permet aux développeurs de créer des applications Web modernes.
COVID-19 à travers le monde
Cette application utilise une visualisation cartographique pour surveiller la propagation du COVID-19, les cas confirmés et le développement de la maladie à travers le monde. Il utilise les données de John Hopkins CSSE.
Traqueur de coronavirus
Il s’agit d’une application Shiny développée par John Coene. Il suit la propagation de COVID-19 en utilisant les données de John Hopkins, les données DXY et Weixin. L’application affiche le nombre de cas suspects, confirmés et récupérés par heure et par région. Le code est disponible sur GithubGenericName.
Cas mondiaux de COVID-19
Cas mondiaux de COVID-19 est une application Shiny développée par Christoph Schoenenberger qui montre les développements de COVID-19 sur une carte, des graphiques, des tableaux récapitulatifs et des chiffres. Son code est disponible sur Github.
Les gouvernements et la COVID-19
Ceci est une application Shiny développée par Sebastian Engel-Loup. Il cartographie la croissance exponentielle du COVID-19, les jours pour doubler les infections, les cas confirmés, le taux de mortalité et le nombre de cas confirmés pour 100 000 personnes dans différentes régions. Le code est disponible sur Github.
Emballer
La communauté open source a réagi rapidement et efficacement à la pandémie de COVID-19. De nombreux projets ont été construits et continuent d’être construits pour lutter contre la propagation de la maladie. Cet article décrit quelques-uns des projets. L’évolution de la maladie dans les semaines à venir reste incertaine. Davantage de projets pouvant tirer parti des technologies open source existantes trouveront leur place dans la lutte contre cette maladie mortelle.
Être prudent!
Article du Dr Michael J. Garbade, PDG, Education Ecosystem